Validation et amélioration d’un algorithme d’identification de poussées de sclérose en plaques dans le SNDS.
Objectifs de l’algorithme
L’algorithme d’identification des poussées de sclérose en plaques (SEP) traitées est un algorithme développé à partir des données médico-administratives du SNDS, chez les sujets atteints de SEP et traités pour la SEP.
L’algorithme identifie les poussées à partir de la combinaison :
- des hospitalisations MCO pour SEP, sélectionnées à partir de codes CIM-10 spécifiques retrouvés dans les diagnostics (principaux, reliés et associés) et de la durée cumulée des séjours associés,
- et/ou des délivrances en ville de corticoïdes à forte dose et de la dose cumulée totale associée.
Auteur(s)
Domaine médical
Méthodologie
L‘algorithme d’identification des poussées de SEP présenté dans ce document a été développé dans le cadre de l’étude EVIDEMS puis validé de manière externe et amélioré dans l’étude DIONISOS.
EVIDEMS a permis de développer un premier algorithme (appelé algorithme initial) sur la base d’avis d’experts et en s’appuyant sur une méthode de validation intra-base. La seconde étude, DIONISOS, est une étude de validation externe de l’algorithme initial qui a permis d’évaluer les performances de l’algorithme initial en le confrontant à un gold standard indépendant et de l’optimiser.
"Méthodologie de l’étude EVIDEMS ayant permis le développement de l’algorithme initial"
L’algorithme a été initialement développé dans le cadre de l'étude EVIDEMS dont l'objectif était d'évaluer l'efficacité du Tecfidera® par rapport aux autres traitements oraux de la SEP (Aubagio®, Gilenya®) et aux traitements immunomodulateurs injectables sur la survenue des poussées au cours du suivi après le début du traitement.
La cohorte de l'étude comprenait tous les patients identifiés dans le SNDS par une première délivrance de traitement modificateur de la SEP (Tecfidera®, Aubagio®, Gilenya® et immunomodulateurs injectables) entre juillet 2015 et décembre 2017, naïf de tout autre traitement de la SEP durant les 4,5 ans précédant l’inclusion, et ayant un suivi compris entre 1 et 3,5 ans (n=9 304). Dans cette étude, le critère d’évaluation principal d’efficacité était le nombre de poussées de SEP survenant au cours du suivi. Une poussée correspond cliniquement à la survenue de nouveaux symptômes ou à la récurrence d’anciens symptômes neurologiques pendant plus de 24 heures. La prise en charge de ces poussées peut se faire soit en ambulatoire, soit en milieu hospitalier et repose sur l’administration de fortes doses de corticoïdes. Les algorithmes d’identification des poussées de SEP étaient jusqu’alors développés uniquement dans les bases de données médico administratives utilisant les codes diagnostics de la Classification Internationale des Maladies 9ème révision (CIM-9). Un algorithme complexe (algorithme initial) a donc été développé dans le SNDS à l’aide d’un comité d’experts, à partir des séquences de délivrances de corticoïdes et des hospitalisations avec un diagnostic compatible avec une poussée de SEP (par exemple, sclérose en plaques, encéphalite, myélite, encéphalomyélite, névrite optique ; codé selon la CIM-10).
Critères d’identification utilisés dans l’algorithme initial

Cet algorithme initial a fait l’objet d’une validation intra-base grâce à une relecture, par des experts cliniciens indépendants, de dossiers médicaux reconstitués à partir des données du SNDS en aveugle des résultats de l’algorithme.
200 patients ont été tirés au sort parmi les patients SEP de la cohorte EVIDEMS :
- 100 patients SEP présentant au moins une poussée pendant la période de suivi détectée par l’algorithme
- 100 patients SEP ne présentant pas de poussée pendant la période de suivi (non détectée par l’algorithme)
100 patients (50 avec au moins une poussée et 50 sans poussée) ont été revus en double aveugle par binôme de 2 experts (chaque binôme comprenait 1 neurologue membre du comité scientifique + 1 neurologue indépendant).
Les cas discordants ont été revus par les experts afin d’ajuster l’algorithme.
L’algorithme a été ajusté afin d’inclure toutes les modifications proposées par les experts, ce qui a permis d’augmenter les performances de l'algorithme : après ajustement, la VPP obtenue est de 95,2% et la VPN est de 100%.
La méthodologie et les résultats sont disponibles dans la publication doi.org/10.1186/s12874-021-01285-y.
Résultat de l’évaluation intra-base (Patients EVIDEMS) :

Langage de programmation
Données utilisées
Données d'application
Le développement de l’algorithme initial a été réalisé sur les données du SNDS entre juillet 2015 et décembre 2018 correspondant à la période de 1 à 3,5 ans de suivi des patients de la cohorte EVIDEMS.
Les données utilisées étaient :
- les tables du DCIR (table des prestations et tables affinées des médicaments) ;
- les tables du PMSI de l’univers MCO (tables des séjours hospitaliers contenant les diagnostics principaux et reliés et table des diagnostics associés).
Validation
L’algorithme initial a été secondairement évalué à partir des données cliniques externes structurées de l’Observatoire Français de la Sclérose en Plaques (OFSEP) chainées via un appariement indirect aux données du SNDS.
Méthodologie de l’étude DIONISOS ayant permis l’évaluation externe de l’algorithme initial et son amélioration.
La cohorte de l’étude comprenait tous les patients atteints de SEP et inscrits dans la base de l’OFSEP entre 2010 et 2019 dont les données ont pu être chainées au SNDS. Parmi les 49 409 patients inscrits dans la base OFSEP et éligibles au chainage avec les données SNDS, 40 051 ont pu être chainés via un appariement indirect, soit un taux d’appariement de 81,1%.
L’étude DIONISOS a été réalisée sur un sous-groupe de ces patients (n=1 640) présentant les mêmes critères d’inclusion que ceux de l’étude EVIDEMS :
- Patients traités pour SEP avec une 1ère délivrance de Tecfidera®, Aubagio®, Gilenya® ou immunomodulateur injectable entre 1er juillet 2015 et 31 décembre 2017 (date index),
- N’ayant aucune délivrance de traitements modificateurs de la SEP (Tecfidera®, Aubagio®, Gilenya® ou traitement immunomodulateur injectable), ni d’autres traitements de la SEP, spécifiques (Tysabri®, Lemtrada®, Elsep®) ou non spécifiques (autres immunosuppresseurs), durant la période historique de 4,5 ans avant la date index.
- Suivis depuis la date index
- jusqu’au 31/12/2018 (si évaluation clinique/épisode neurologique dans l’OFSEP après et pas de décès avant
- ou toute dernière date d’évaluation clinique / épisode neurologique dans l’OFSEP (si ≤ 31/12/2018)
- ou date de décès (si ≤ 31/12/2018)
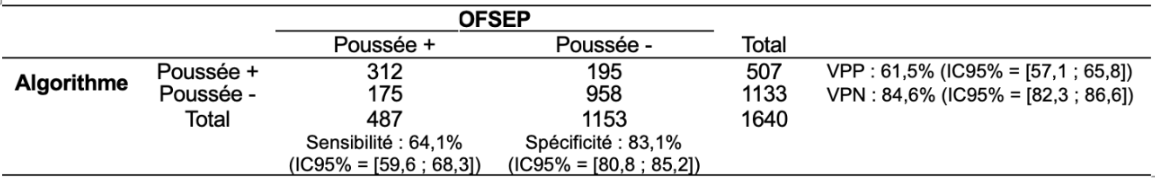
L’évaluation de l’algorithme initial a tout d’abord été réalisée sur les 1 640 patients (population principale).
La première poussée de SEP de chaque patient a été identifiée par l’algorithme au cours du suivi dans les données du SNDS, puis a été comparée aux données de poussées identifiées dans l’OFSEP. Les performances de l’algorithme sont modérées avec une VPP modérée et une bonne VPN.
Résultats de l’évaluation externe (Population principale) :
Suite à cette évaluation, des modifications ont été apportées à l’algorithme grâce à l’appui d’experts de la SEP concernant :
- Élargissement des codes diagnostics hospitaliers compatibles avec une poussée de SEP (codes CIM-10)
- Ajout des codes GHM spécifiques de SEP
- Ajout des codes GHM spécifiques de chimiothérapie
- Suppression de la borne supérieure de la dose de corticothérapie
- Suppression de la borne supérieure de la durée d’hospitalisation
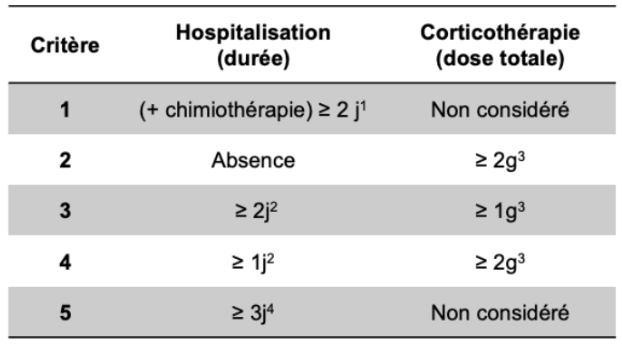
Critères d’identification utilisés par l’algorithme optimisé :
Ces modifications ont permis d’améliorer les performances diagnostiques de l’algorithme qui se trouvent désormais meilleure pour la VPP et très bonne pour la VPN.
Résultats de l’amélioration externe (Population principale) :

Finalement, l’algorithme optimisé a été testé dans une population élargie à tous les patients présentant ou non une délivrance de traitements de la SEP, spécifiques (Tysabri®, Lemtrada®, Elsep®) ou non spécifiques (autres immunosuppresseurs), durant la période historique (n=9 966). Les performances de l’algorithme dans cette population sont modérées, présentant une faible VPP (55,3% (IC95% = [53,6 ; 57,1]) et une bonne VPN (91,6 (IC95% = [90,9 ; 92,3]). La diminution des performances dans cette population est probablement liée à l’hétérogénéité de la population incluant des personnes non traitées avant la prise d’un traitement modulateur et de personnes traitées pour leur SEP depuis plus ou moins longtemps. L’hétérogénéité des niveaux de sévérité doit se refléter dans les critères de prise en charge permettant de repérer une poussée de SEP dans le SNDS.
L’algorithme optimisé peut donc être utilisé avec un bon niveau de confiance chez les patients atteints de SEP et initiant un traitement modificateur de la SEP (traitements oraux (Tecfidera®, Aubagio®, Gilenya®) et traitements immunomodulateurs injectables), naïfs de tout autre traitement de la SEP.
Date de dernière mise à jour
Juin 2024
Maintenance
Pas de maintenance de l’algorithme
Comment installer l’algorithme ?
Il s’agit d’une macro SAS nommée %RLPSEPSNDS, exécutable sous SAS, permettant de générer une table de données SAS contenant des informations (dates, mode d’identification) sur les poussées SEP identifiées.
Un prétraitement des données est requis (voir section 2.10 / Étape 1).
Comment utiliser l’algorithme ?
Pour obtenir les résultats de l’algorithme d’identification des poussées de SEP dans le SNDS, effectuer les étapes suivantes :
Étape 1 : Préparation des données
3 tables sont requises dans l’exécution des programmes et sont donc à créer avant de lancer la macro :
- Table « PATIENTS » (1 ligne/patient) de l’étude avec un identifiant unique : ID_PATIENT
- Table « MEDICAMENTS » (1 ligne/médicament/date du médicament/patient) avec les variables suivantes :
- ID_PATIENT (Identifiant unique) ;
- EXE_SOI_DTD (date de délivrance, format date) ;
- PRE_PRE_DTD (date de prescription, format date) ;
- NOM_MEDIC (nom du traitement d’intérêt : SOLUMEDROL ou MEDROL, type alpha-numérique) ;
- DOSAGE_MEDIC (dosage en mg : 1000, 100 ou 120, type numérique) ;
- UNITE_MEDIC (nombre d’unités par boîte : 1 ou 30, type numérique) ;
- PHA_ACT_QSN (nombre de boites délivrées, type numérique) ;
La table MEDICAMENTS doit être restreinte aux traitements d’intérêt suivants :
- Medrol 100 mg (methylprednisolone) 1 boite de 30 ;
- Solumedrol 1000 mg (methylprednisolone) 1 boite de 1 ;
- Solumedrol 120 mg/2 ml (hemisuccinate de methylprednisolone) 1 boite de 1
Cette table est à générer à partir des données suivantes et sera restreinte aux ID_PATIENT de la table « PATIENTS » :
- Tables issues du DCIR : ER_PRS_F / ER_PHA_F / ER_UCD_F ;
- Le référentiel de la pharmacie IR_PHA_R complété éventuellement par d’autres sources de données des médicaments pour limiter les données manquantes (code ATC, dose, nombre d’unités).
- Table « HOSPITALISATIONS » (1 ligne/séjour hospitalier/patient : hospitalisation de jour (HDJ) ou séjour ≥ 1 nuitée) avec les variables suivantes :
- ID_PATIENT (identifiant unique) ;
- ETA_NUM (identifiant séjour, type alpha-numérique) ;
- RSA_NUM (identifiant séjour, type alpha-numérique) ;
- GRG_GHM (code GHM du séjour, type alpha-numérique) ;
- EXE_SOI_DTD (date de début de l’hospitalisation, format date) ;
- EXE_SOI_DTF (date de fin de l’hospitalisation, format date) ;
- DP1 DP2 DP3 … DPn1* (diagnostics principaux en colonnes par séjour, type alpha-numérique) ;
- DR1 DR2 DR3 … DRn2* (diagnostics reliés en colonnes par séjour, type alpha-numérique) ;
- DAS1 DAS2 DAS3 … DASn3* (diagnostics associés en colonnes par séjour, type alpha-numérique) ;
* n1, n2 et n3 varient selon le nombre des diagnostics (DP/DR/DAS) retrouvés dans chaque séjour.
Le code CIM-10 ne doit pas contenir : "." "," "+" ou autres caractères que des chiffres et des lettres.
La table est à générer à partir des données SNDS MCO suivantes et sera restreinte aux ID_PATIENT de la table « PATIENTS » : T_MCOxx_B / T_MCOxx_UM / T_MCOxx_D / T_MCOxx_C.
Étape 2 : Exécution de la macro
La macro %RLPSEPSNDS(Entree=, Sortie=) peut maintenant être exécutée. Les paramètres à renseigner sont les suivants :
- Entree : nom de la librairie SAS contenant les 3 tables générées à l’étape 1 (les noms des tables et des variables cités ci-dessus doivent être respectés). Ce paramètre doit obligatoirement être renseigné ;
- Sortie : nom de la librairie SAS recevant la table des résultats.
Étape 3 : Consultation et vérification des résultats
Une table finale nommée Poussees_SEP_SNDS (1 ligne/poussée/patient) est générée dans la librairie « Sortie »avec les variables suivantes :
- ID_PATIENT (identifiant unique);
- Critères (codée de 1 à 5 (type numérique) et correspondant aux 5 critères d’identification des poussées de SEP utilisés par l’algorithme: 1 et 5 - identification par l’hospitalisation uniquement,
2 - identification par le traitement par corticothérapie uniquement,
3 et 4 - identification par l’hospitalisation + corticothérapie) ;
- EXE_SOI_DTD (date de début du critère, format date) ;
- EXE_SOI_DTF (date de fin du critère, format date).
Remarques :
- la macro s’exécute sur toutes les données disponibles et valides ;
- l’indicateur « criteres » correspond aux poussées identifiées (une poussée = un critère) ;
- l’algorithme considère 2 poussées comme identiques si elles se suivent dans un délai de 31 jours (les poussées séparées de moins de 31 jours ne sont comptabilisées qu’une seule fois) ;
- l’algorithme ne considère pas comme une nouvelle poussée les répétitions régulières des poussées SEP identifiées (si au moins 3 poussées distinctes sont identifiées, espacées d’au moins 40 jours (+/- 15 jours) et de moins de 90 jours (+/- 15 jours) alors on ne considère que la première). En effet, il est peu probable que des hospitalisations et/ou des délivrances de corticoïdes répétées voire régulières sur une courte durée correspondent à des poussées distinctes.